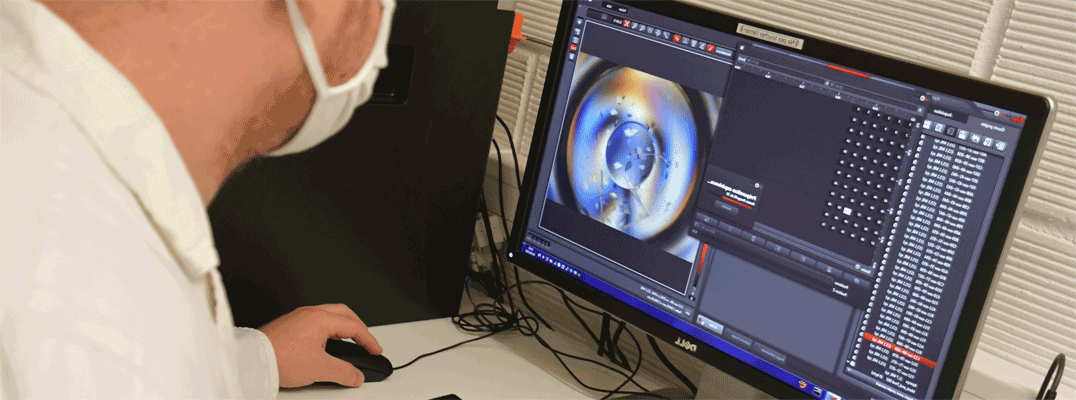
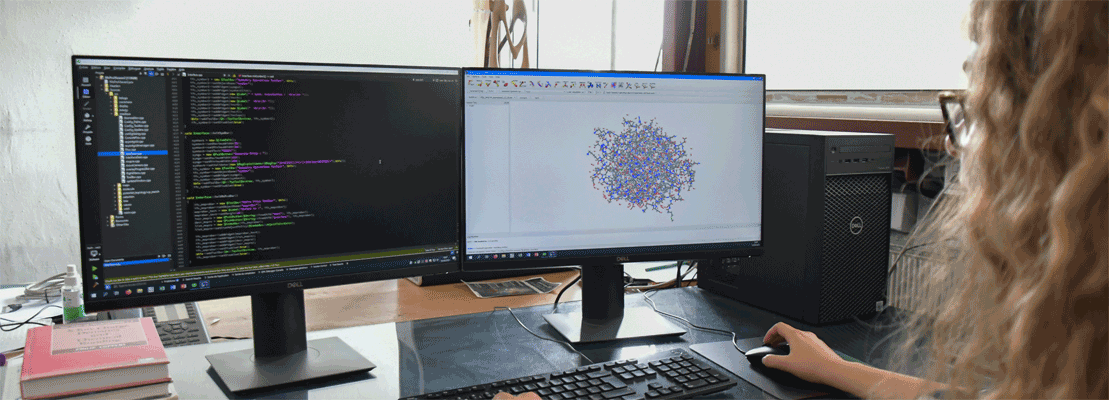
En savoir plus…
Auteurs : Christian Jelsch, Benoit Guillot, Niels Hansen, Claude Lecomte, Virginie Pichon-Pesme
MoPro est un programme cristallographique dédié à :
- l’affinement de structure moléculaires et de leur densité électronique à résolution subatomique ( d ~ 0,4 – 0,7Å)
- l’affinement de structure à résolution atomique ( d ~ 0,7 – 1,5Å) en utilisant un modèle multipolaire de l’atome transféré depuis une librairie de densité de charge.
Les domaines d’application sont :
- les molécules organiques et organométalliques, les cristaux de minéraux,
- les macromolécules biologiques (protéines, ADN, ARN)
Le programme permet l’application de Restrictions et Contraintes originales à la structure moléculaire et à la densité électronique. MoPro inclut des fonctionnalités d’analyse de la stéreo-chimie et de la dynamique moléculaire. En exportation, MOPro peut écrire notamment des fichiers de coordonnées en format CIF et PDB en vue de publication.
Import2MoPro
Programme interactif qui lit les fichiers moléculaires de type PDB, CIF, MOLLY & SHELXL.
VMoPro
Visualisation des propriétés moléculaires dérivées en vue 2D ou 3D.
- Densité Electronique Statique : densité totale ou de déformation
- Densité Electronique Dynamique par Synthèse de Fourier
déformation de la densité : [ Fobs(mul) – Fcalc(sph) ]
densité électronique résiduelle [ Fobs – Fcalc ]
ou plus généralement : [ mFo – nFc ]
- Potentiel Electrostatique
- Gradient & Laplacien de la densité électronique. Points critiques
- Calculs d’Energie d’Interactions Moléculaires
En savoir plus…
Auteur : Benoit Guillot
MoProViewer est un visualisateur moléculaire dédié au domaine de l’analyse de la densité de charge, lié à la suite de programmes de raffinement MoPro et plus particulièrement au programme VMoPro, qui calcule les propriétés dérivées de la densité électronique. MoProViewer peut être considéré comme une interface graphique de VMoPro car il permet de configurer graphiquement un calcul et d’afficher les résultats. MoProViewer peut afficher des isosurfaces 3D, des cartes d’isocontours 2D, des points critiques et des chemins de liaison, des moments électrostatiques, des champs de lignes de gradient et des bassins topologiques.

En savoir plus…
Auteur : Massimo NESPOLO
Publication :
M. Nespolo, C. Iordache (2013). « Twiny: from morphology to twin element and vice versa » – Journal of Applied Crystallography 46 (2013) 801-803. Electronic reprint (354 Ko) (DOI)
Electronic reprint (354 Ko) (DOI)
Le logiciel Twiny calcule l’angle entre deux cristaux maclés à partir de l’élément de macle ou estime l’élément de macle à partir de l’angle mesuré entre deux cristaux maclés.
Fichier Windows : Twiny-win.zip (6 Mo)
Fichiers Linux : 32bits / 64bits (6 Mo)
Fichier Mac : Twiny-mac.zip (25 Mo)
En savoir plus…
Auteur : Massimo NESPOLO
Publications :
- M. Nespolo (2016). Charge Distribution as a tool to investigate structural details. IV. A new route to heteroligand polyhedra. Acta Crystallogr., section B, in press.
- M. Nespolo, B. Guillot (2015). CHARDI2015: Charge Distribution analysis of non-molecular structures. J. Appl. Crystallogr., submitted
Le logiciel CHARDI2015 calcule le Nombre de Coordination Effective (ECoN) et la Distribution de Charges (CD) Charge Distribution (distribution des nombres d’oxydation formelles) dans les composés à valence normale (NVC). Les NVC sont composés inorganiques sans liaisons cation – cation ou anion – anion. ECoN est un nombre de coordination fractionnaire qui mesure la distorsion des polymères de coordination, et qui coïncide avec le nombre de coordination classique lors que cette distorsion est limitée. Les « charges » des atomes sont utilisées comme poids fans la distribution d’CoN parmi les liaisons chimiques, dans le cadre d’un modèle à charges ponctuelles. Les résultats sont utilisés pour la validation structurelle et pour l’analyse des détails structuraux.
Fichier Windows : Chardi2015.zip (9 Mo)
En savoir plus…
Auteur : Massimo NESPOLO
Publication :
M. Nespolo, H. Takeda, T. Kogure, G. Ferraris (1999). Periodic Intensity Distribution (PID) of mica polytypes: symbols, structural model orientation and axial settings. Acta Crystallogr. A55, 659-676
Le logiciel PTST98 calcule la fonction Distribution Périodique d’intensité (PID) comme transformé de Fourier de la séquence d’empilement de feuillets mica exprimée par les symboles Ross-Takeda-Wones (RTW). Le résulté est comparé avec la PID expérimentale, obtenue en supprimant l’effet de modulation de la transformé de Fourier du feuillet sur les facteurs de structure du polytype. Le meilleur accord entre la PID théorique et celle expérimentale révèle la séquence d’empilement.
Fichier Windows : PTST.zip (379 Ko)
En savoir plus…
Auteur : Massimo NESPOLO
Publication :
M. Nespolo, G. Ferraris (2006). The derivation of twin laws in non-merohedric twins – Application to the analysis of hybrid twins. Acta Crystallogr. A62, 336-349. Electronic reprint (330 ko)
Electronic reprint (330 ko)
Le logiciel geminography explore les possibles lois de macles. Il prend en input les paramètres de maille et les indices de l’élément de macle (connu ou soupçonné), plus d’autres paramètres décrits dans la documentation, et explore les éléments du réseau (quasi)-perpendiculaires pour repérer les possibles sous-réseaux. Il calcule l’indice de macle et l’obliquité, ainsi que la pseudo-symétrie des sous-réseaux. Ce logiciel effectue une recherche systématique des sous-réseaux coexistant et décrit les macles comme hybrides chaque fois que cette description rend compte de la quasi- superposition réticulaire mieux que la description classique, qui utilise au contraire un seul sous-réseau.
Fichier Windows : Install_geminography.zip (6 Mo)